《自然》重磅专题:9篇论文齐发!全面展示迄今最详细的小鼠全脑细胞图谱
发布时间:2023-12-182023年12月13日,顶尖学术期刊《自然》的最新一期如期上线,同时发布了美国国立卫生研究院脑科学计划-细胞普查联盟(BICCN)的9篇论文,展示了科学家们创建小鼠全脑高分辨率细胞图谱的最新成果。
《自然》介绍,这组研究成果为小鼠完整大脑的细胞类型提供了迄今为止最全面、最详细的特性描述和分类,不仅有数千种细胞类型的大量信息,还介绍了这些细胞如何连接,每个细胞中活跃的基因和调控基因的程序,以及基因调控如何驱动细胞功能。这些工作奠定了一个坚实的基础,可供进一步研究哺乳动物大脑的发育和演化,并为神经生物学和神经系统疾病的新发现提供重要工具。
前沿技术揭示空间位置的重要性
与过去探测大脑的工作相比,这份新图谱不再受限于某个选定的区域,而是对小鼠整个大脑的详细调查,因此不仅揭示了以往研究不足的脑区有令人惊讶的细胞多样性,也可以更深入地理解各种神经回路的组织和功能,从而理解大脑的运作。

在该专题的旗舰论文中,艾伦研究所(Allen Institute)的曾红葵博士等研究人员描述了首个完整小鼠大脑高分辨率转录组细胞类型图谱的创建方法以及主要发现。
研究团队结合了两个海量数据集——约700万个细胞的单细胞RNA测序(scRNA-seq)数据和约430万个细胞的空间转录组学(MERFISH)数据,将小鼠全脑细胞分为34个大类(class),再进一步细分为338个子类(subclass),1201个超型(supertype),5322个细胞集群(cluster)。其中,神经元的细胞类型显示出惊人的多样性,共有5205个集群。
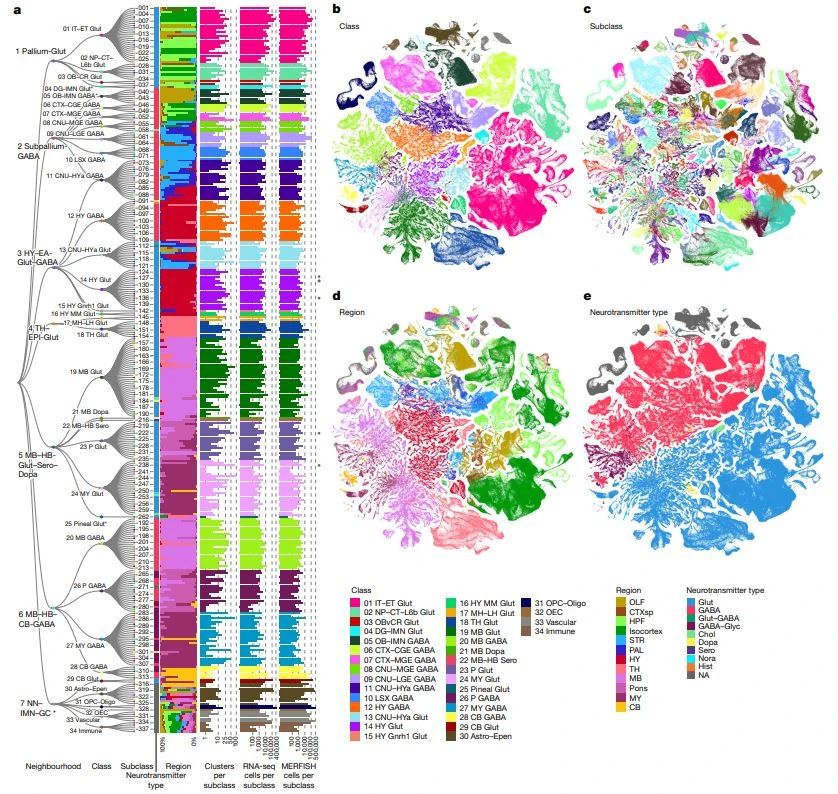
▲小鼠全脑根据转录组特征定义的细胞类型(图片来源:参考资料[1])
研究人员强调,空间分辨率高是这份图谱的独特之处,因此可以将每种细胞的转录组特征与其空间位置高度对应,从中清楚地看到大脑解剖结构与细胞类型之间的关系。
例如,大脑的背侧和腹侧,细胞类型的组织存在明显差异:背侧区域含有的细胞类型较少,但高度分化;腹侧则包含了更多类型的神经元,但类型之间的关系较近。这种差异与不同结构的功能和演化有关。背侧部分包括皮质、丘脑和小脑等,主要执行生物体的适应性功能,如认知、感觉运动等,演化时间较晚,细胞类型在多样性上的拓展速度较快;腹侧包括下丘脑、中脑和后脑等较古老的结构,承担了进食、繁殖、新陈代谢平衡等更基本的生存功能,专用细胞类型和神经回路的变化就相对较小。

由麻省理工学院和哈佛大学博德研究所的科学家领导的第二篇论文,采用了不同但互补的方法,将新开发的空间转录组学技术Slide-seq与高通量单核RNA测序相结合,揭示了小鼠全脑各个解剖结构中的细胞类型组成,包括过去关注较少的中脑、脑桥、延髓和下丘脑。研究人员估计,分析已涵盖小鼠大脑中约90%的细胞类型。研究团队通过在线资源的形式提供了这些数据:www.BrainCellData.org。
▲研究人员分析了单个小鼠大脑的101个slide-seq切片,从而深入全面了解整个大脑的细胞类型。这张可视化图中切片被堆叠在一起,以说明小鼠大脑已知主要区域的覆盖情况,不同区域以不同颜色标出。(图片来源/Credit:Macosko&Chen实验室)
在这两项研究中,科学家们还全面描述了大脑中的信号分子——神经递质和神经肽的特征,包括它们在不同脑区的不同细胞类型中的表达和共表达模式,同样显现出丰富的多样性和异质性。
除了各种神经元外,非神经元类型的细胞如星形胶质细胞、外胚层细胞、伸展细胞、血管软脑膜细胞等,也具有非常明显的空间特异性,表明了神经元与胶质细胞、胶质细胞与血管之间的特异性相互作用。
此外,研究确认了转录因子具有决定整个大脑细胞类型分类的作用。研究人员列出了整个大脑各个部位决定每一种细胞类型的主要转录因子组合。

哈佛大学庄小威实验室开发的多重容错性荧光原位杂交(multiplexed error-robust fluorescence in situ hybridization,简称MERFISH)技术在图谱的创建中发挥了重要作用。这项技术可以让研究人员在细胞或组织切片中同时检测多种RNA,从而绘制选定区域的基因表达空间图谱。
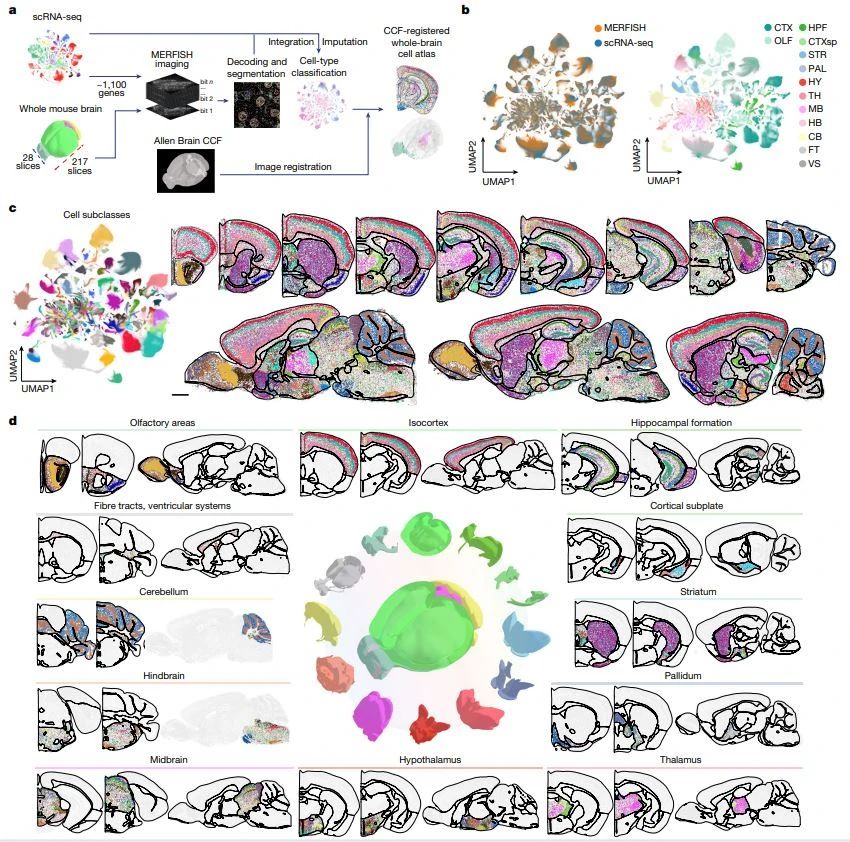
▲基于MERFISH构建高分辨率小鼠全脑细胞图谱(图片来源:参考资料[3])
此次,庄小威研究团队将MERFISH与单细胞RNA测序数据结合,对成年小鼠全脑约1000万个细胞中的1100多个基因进行成像,放入艾伦研究所的小鼠全脑通用坐标框架中,创建出单细胞空间图谱(每个细胞都有全转录组表达谱),可以系统地定量分析各个脑区的细胞类型和组成,推断细胞子类之间的特异性相互作用,预测配体-受体之间的分子关系,以及细胞-细胞相互作用的功能。
基因调控
另外四篇论文中,多个研究团队应用小鼠全脑转录组细胞类型图谱,分析了不同细胞类型的基因调控程序,并用于识别造成神经系统疾病和特性的遗传变异。

大脑中的许多细胞类型通过远距离投射来调节细胞功能,还有许多特定细胞类型之间具有相互作用。索尔克研究所的科研团队将单细胞表观基因组、细胞类型与小鼠各脑区域的神经元长距离投射联系起来。
这个数据集涉及大脑32个不同区域,以及超过3万个大脑神经元。借助该数据集,研究者可以量化投射到不同目标脑区神经元之间的遗传差异,对分子细胞类型和它们的投射靶标进行注释,并且在投射富集的细胞类型中构建基因调控网络。

胞嘧啶DNA甲基化对大脑发育非常关键,并与各种神经系统疾病有关。因此,另一篇研究尝试在空间背景下理解大脑的DNA甲基多样化。
借助单核甲基化组测序以及多组学测序技术,作者从成年小鼠大脑的117个解剖区域获得了超30万个甲基化组和超17万个染色质构象/甲基化组联合谱。以此为基础,研究构建了一个基于甲基化的细胞分类系统,包括4673个细胞群和274个跨膜态注释亚群,这些都代表了潜在的基因调控元件。
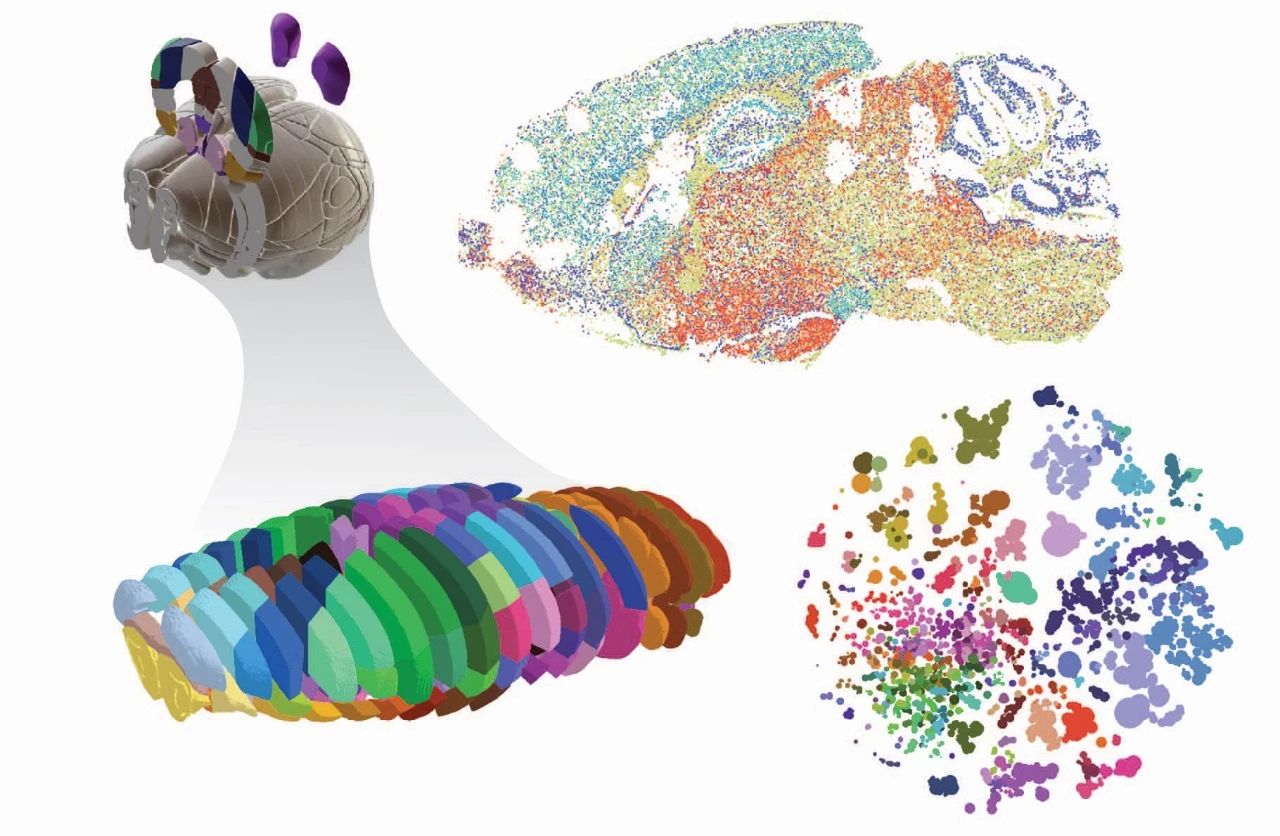
▲左上-基于解剖脑区的小鼠大脑的3D效果图;左下-不同脑区标志为不同颜色的小鼠大脑3D效果图;右上右下-小鼠大脑的垂直切片,不同颜色代表不同细胞类型,圆圈代表根据表观基因组分析在小鼠大脑中发现的细胞类型的数量和多样性。(图片来源/Credit:索尔克研究所)
研究还在大脑区域内和跨区域的细胞类型中观察到了基因和调控元件的胞嘧啶甲基化模式。这些全脑单细胞DNA甲基化组图谱可以为了解小鼠大脑细胞空间、基因组调控多样性提供大量资源。
在上述两篇论文中均担任共同通讯作者的Joseph Ecker教授指出:“当你利用这个基于表观基因组的细胞图谱,开始研究已知会导致人类疾病的基因变异时,你就会对哪些细胞类型在疾病中最容易受到伤害有新的认识。”

另一篇文章中,加利福尼亚大学圣地亚哥分校的任兵教授领导团队绘制了成年小鼠大脑中顺式调控DNA元件(cCRE)的综合图谱,该图谱通过分析230万个脑细胞的染色质可及性生成。整个图谱包括约100万个cCRE,以及它们在不同脑细胞群中的染色质可及性。基于此,新研究为小鼠基因组新增加了44.6万多个cCRE。
根据图谱,作者发现转座子在新的调控程序和神经元多样性中具有潜在作用。研究还以此建立了深度学习模型,可以仅从DNA序列就预测不同脑细胞类型中基因调控元件的活性。

任兵教授团队主导的另一篇论文,利用单细胞多组学方法研究了人类、猕猴、小鼠初级运动皮层的基因调控程序,从超过20万个细胞中获得了基因表达、染色质可及性、DNA甲基组和染色体构象图谱。
数据显示,转录因子表达差异与物种特异性表观基因组相对应。在大脑皮质细胞中,转座因子占据了人类特异性顺式调控元件的约80%。
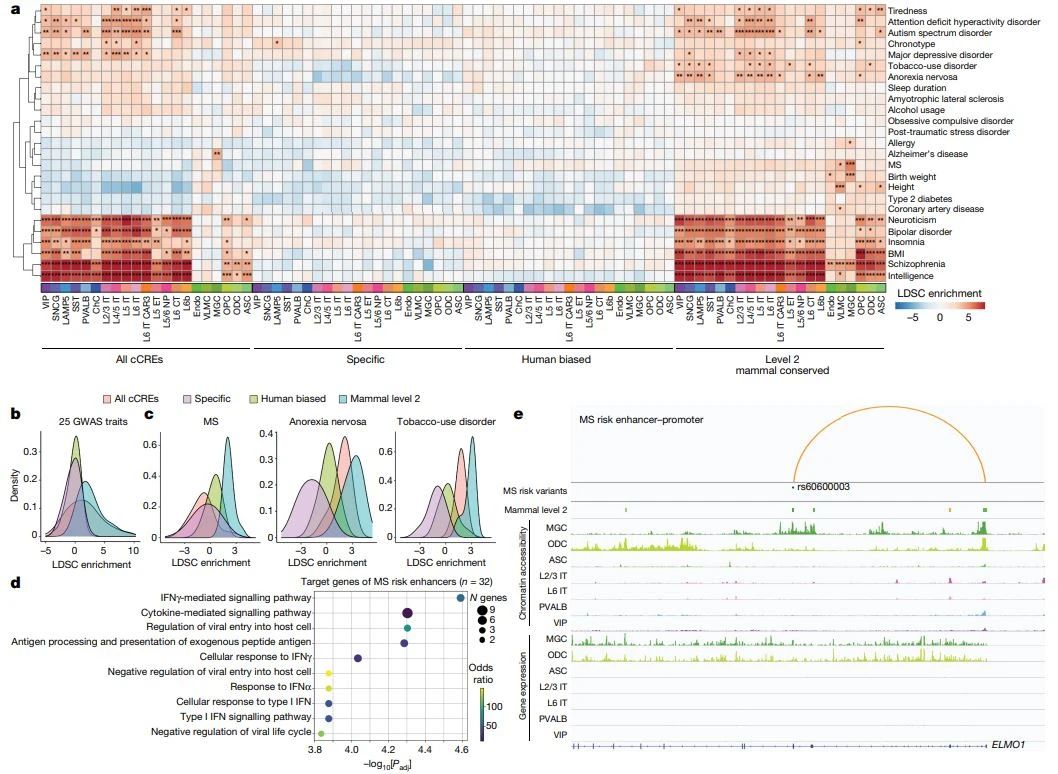
▲利用表观遗传学保守特征解释与神经系统疾病风险相关的非编码变异(图片来源:参考资料[7])
通过机器学习,研究在不同物种中开发了可以预测顺式调控元件的预测因子,并且确认从啮齿动物到灵长类动物,基因组的调控规则得到了高度保守。这项工作还发现了与多发性硬化、厌食和烟瘾相关的遗传变异中的保守特征。
理解大脑演化的新工具
《自然》同时刊发的“新闻与观点”文章中,专家指出:“细胞类型图谱的重要性,不仅在于在细胞尺度上理解大脑结构,也在于对大脑演化做出准确推断。”
此次的一篇论文中,加州大学伯克利分校的研究团队就展示了利用细胞类型图谱窥视动物眼睛的演化之谜。

视网膜是眼睛直接与脑部相连的结构,也是视觉感知的起点。不同动物的视觉差异巨大,但新研究却指出,不同脊椎动物视网膜的细胞类型有着惊人的一致性。
作者首先生成并比较了包括人类在内17种脊椎动物的视网膜单细胞转录组图谱,这些物种跨越了广泛的动物类型,但6种视网膜细胞类型(光感受器、水平细胞、双极细胞、无长突细胞、视网膜神经节细胞和穆勒胶质细胞)有着高度分子保守性。基于保守的基因表达程序,众多细胞类型在不同物种间共享。
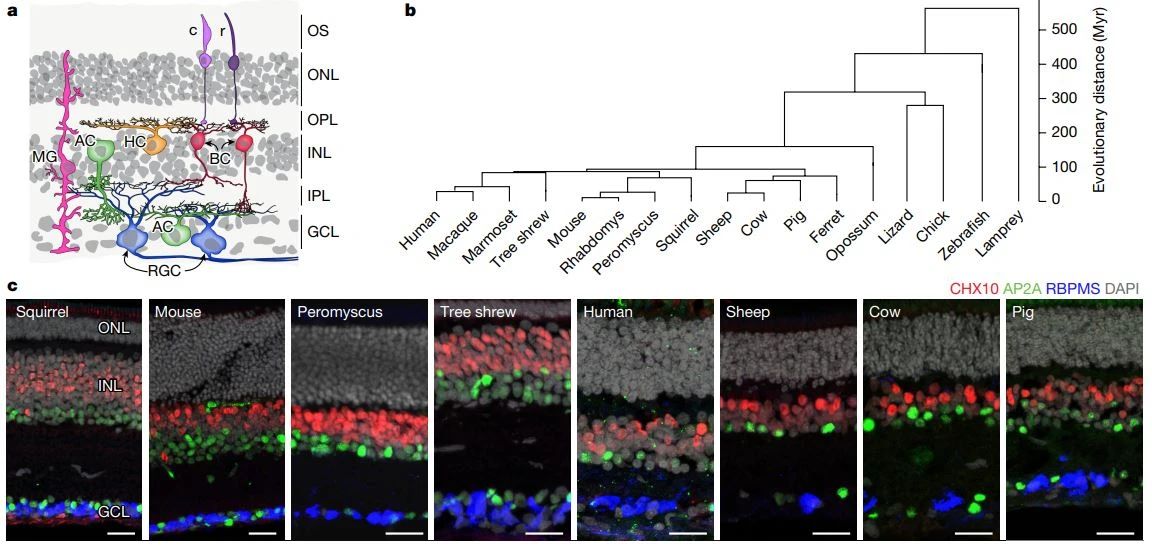
▲脊椎动物的视网膜具有演化上保守的基本细胞结构(图片来源:参考资料[1])
这些发现指向了一个结论:早在2亿年前,所有哺乳动物最后共同祖先的视网膜,就已经具有与现代哺乳动物相媲美的复杂性;其中一些细胞类型的历史,甚至可以追溯到4亿多年前所有脊椎动物的共同祖先。
这项研究还为一类视网膜神经节细胞(RGC)的起源提供了全新的认知。侏儒视网膜神经节细胞(midget RGC)占据了人类RGC细胞的80%以上,此前被认为源于灵长类动物,它们为人类高度灵敏的视力提供了基础。但基于对小鼠脑图谱的对比研究发现,小鼠视网膜中也有与侏儒RGC同源的细胞。与人类的侏儒RGC相比,小鼠的这种细胞体型更大但数量更少,仅占小鼠RGC的2%~4%。在演化过程中,这类细胞体型可能变小、同时数量增加,这些变化与视觉皮层的扩大平行发生。这些发现也为我们探索眼部疾病机制提供了重要线索。
参考资料:
[1] A high-resolution transcriptomic and spatial atlas of cell types in the whole mouse brain
Nature. (2023) DOI: 10.1038/s41586-023-06812-z R
[2] Langlieb J, Sachdev NS, et al. The cell type composition of the adult mouse brain revealed by single cell and spatial genomics. Nature. (2023) DOI: 10.1038/s41586-019- 0000-0
[3] Meng Zhang et al., Molecularly defined and spatially resolved cell atlas of the whole mouse brain. Nature. (2023) DOI: 10.1038/s41586-023-06808-9
[4] Jingtian Zhou et al., Brain-wide correspondence of neuronal epigenomics and distant projections. Nature. (2023) DOI: 10.1038/s41586-023-06823-w
[5] Hanqing Liu et al., Single-cell DNA methylome and 3D multi-omic atlas of the adult mouse brain. Nature. (2023) DOI: 10.1038/s41586-023-06805-y
[6] Songpeng Zu et al., Single-cell analysis of chromatin accessibility in the adult mouse brain. Nature. (2023) DOI: 10.1038/s41586-023-06824-9
[7] Nathan R. Zemke et al., Conserved and divergent gene regulatory programs of the mammalian neocortex. Nature. (2023) DOI: 10.1038/s41586-023-06819-6
[8] Carla C. Winter et al., A transcriptomic taxonomy of mouse brain-wide spinal projecting neurons. Nature. (2023) DOI: 10.1038/s41586-023-06817-8
[9] Joshua Hahn et al., Joshua Hahn et al., Evolution of neuronal cell classes and types in the vertebrate retina. Nature. (2023) 10.1038/s41586-023-06638-9
来源:学术经纬

